マルチステップ=マルチスケール・ブートストラップ法
- 確率値の計算
- 高精度リサンプリング法の開発
- バイオインフォマティクスへの応用
- 参考資料
1. 確率値の計算
近年ゲノム科学など様々な分野で膨大なデータが急速に蓄積されています.これから知識発見を行うために,データマイニングなどのデータ解析手法によって非常に多くの仮説が同時に探索されることがあります.このような状況では,データに内在する確率的な揺らぎ,すなわちバラツキの影響が増幅されて誤った発見に導かれやすくなるので,この影響を正しく評価することが重要になります.
リサンプリング法はデータからのサンプリングによってバラツキを評価する一般的な手法であり,一種の確率シミュレーション技法です.代表的なリサンプリング法であるブートストラップ法 (Efron 1979)が発表されて20年以上が経ち,様々なデータ解析に利用されていますが,その精度が必ずしも十分でないことが分かってきました.より精度の高い方法が必要です.
現実のデータ解析は手続きが複雑で出力も多様です.そこで広範な応用を可能にするために,興味のある仮説を支持したか否かという二者択一の結果だけを計算に利用する方法を考えます.ブートストラップ法によって生成した多数の複製データが仮説を支持した頻度はブートストラップ確率と呼ばれ,バイオインフォマティクスの応用で以前から利用されています (Felsenstein 1985).ブートストラップ確率は,仮説検定の確率値として解釈できます.
2. 高精度リサンプリング法の開発
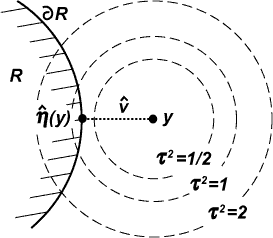
不偏な確率値は母数空間における幾何学によって表現されます.すなわち,データ点から仮説までの「符号付距離」や仮説の「曲率」などの幾何的量を用いると,3次の精度の近似的に不偏な確率値の式が書けます.一般にk 次の精度の確率値とは,サンプルサイズn の増加にしたがい検定のバイアスがn-k/2 に比例して小さくなることを意味します.高精度リサンプリング法の一種として新たに開発されたマルチスケール・ブートストラップ法では,ブートストラップ複製のサンプルサイズを変えて複数回(2回以上)のブートストラップ法を実行し,そのときのブートストラップ確率の変化率から幾何的量を推定します(図1).データが何らかの滑らかな非線形変換によって正規モデルになること仮定すれば,任意の滑らかな境界をもつ仮説に対して,この方法は3次の精度の確率値を計算します.通常のブートストラップ法から計算したブートストラップ確率は一般に1次の精度ですから,それよりも高精度確率値が計算できることを意味します.
さらに,2ステップ=マルチスケール・ブートストラップ法から,「加速定数」(確率モデルの母数空間のゆがみを表す量)も2次の精度で推定できるので,データが何らかの指数型分布族に変換できることを仮定すれば,2次の精度の確率値が計算できます.3ステップ法では3次の確率値になります.いずれの方法も,ダブルブートストラップ法のようなネストのブートストラップ法ではなく,通常のブートストラップ法の定数倍の計算量で実行できます.背後にある理論はかなり煩雑な計算を伴うので,数式処理ソフト (MathematicaとMathTensor) を用いて数学的な証明を行っています.ところが得られたアルゴリズムはとてもシンプルで実装も容易です.
|
 |
|
|
|
 |
|
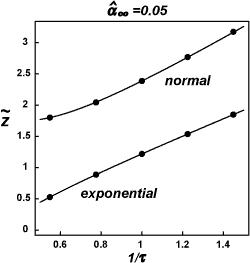
| 図1.(左) ブートストラップ法の幾何的解釈.ブートストラップ複製のバラツキを点線で表現してある.ブートストラップ確率は複製が領域Rに入る頻度である.スケールτ を変化させるとバラツキの大きさが変わる.(右) スケールを変化させたときのブートストラップ確率の変化.横軸はスケールの逆数.縦軸はブートストラップ確率のz値.このグラフの曲線のτ=1における傾きが不偏な確率値のz値になる.詳細はShimodaira (2004a)を参照. |
|
|
|
|
|
|
3. バイオインフォマティクスへの応用
生物のDNAを比較して進化の系統樹を推定する問題(「確率モデルで探る生物の進化」を参照)に,マルチスケール・ブートストラップ法を応用した実例を紹介します(詳細はShimodaira (2002)を参照).哺乳類のミトコンドリアDNAを比較すると,進化の過程で生物の共通祖先からどのような順序で枝分かれしたかが推測できます.ここでは5個の哺乳類グループの比較を行い,1=ヒト,2=(アザラシ,ウシ),3=ウサギ,4=マウス,5=オポッサムとラベル付けします.可能な系統樹の組み合わせを数えると,1 x 3 x 5 = 15とおりであることが分かります(図2).つまり真実の系統樹はこの15通りのどれかひとつであると考えられます.
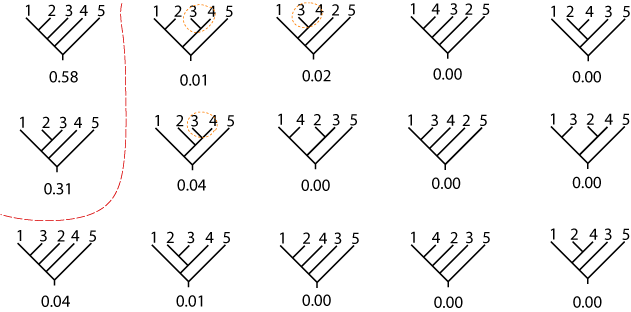
図2.ブートストラップ確率.
まず通常のブートストラップ法を適用してブートストラップ確率を計算した結果が図2の各系統樹の下に書いてあります.これがある閾値以上の系統樹は真実である可能性があると判断し,逆にそれより小さい系統樹は真実ではないと判断します.閾値として一般に良く用いられる0.05を使うと,赤線で囲んだ2個の系統樹が選ばれます.残りの13個は真実でないと判断されます.
古典的な形態学によって推測された系統樹とこの結果が矛盾するとしてNature誌等に論文が掲載されました.形態学ではウサギとマウスが近縁であり,図の3番と4番の枝がひとつのグループになると考えられていました(オレンジ色の点線).ところがこの古典的な仮説に対応した3個の系統樹は真実でないと判断されたので,従来の仮説と矛盾するような生物学的に新しい発見をしたとみなされたのです.
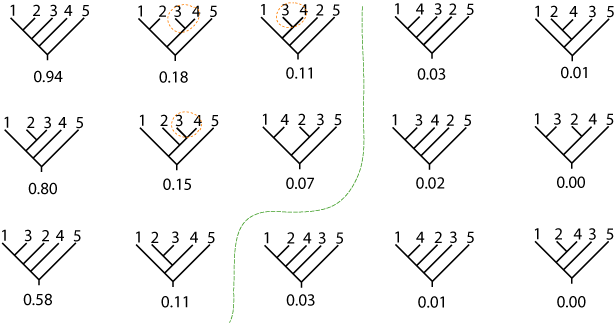
図3.下平・長谷川検定による確率値.
一般にブートストラップ確率は精度が低く,系統樹推定に応用すると,あやまった発見に結びつく可能性が高いことが分かっています (false positiveの確率が大きい).そこで,この誤り確率を小さくする手法を開発して(下平 1993, Shimodaira 1998),系統樹推定に応用しました (Shimodaira and Hasegawa 1999,被引用回数).この方法は統計学で古くから知られている多重比較法という考え方をモデル選択に応用したものであり,現在では下平・長谷川検定(Shimodaira-Hasegawa test)と呼ばれ,分子進化学の標準手法の一つとして広く利用されています.動植物の進化の解明にとどまらず,HIVやSARSウイルスの研究でも重要な役割を果たしています.
計算した確率値を図3に示します.確率値が閾値0.05以上になる系統樹は緑線で囲んだ8個あり,ウサギとマウスが近縁であるという古典的な仮説も真実の可能性があると判断されます.つまり生物学的な発見をしたとはみなされないことになります.その後得られた核DNAのデータも含めて再分析したところ,どうやら図3の7番目の系統樹(確率値が0.11のもの)が真実である可能性が高いと現在では考えられています.
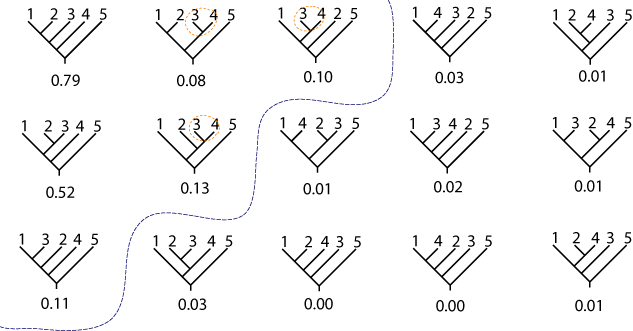
図4.マルチスケール・ブートストラップ法による確率値.
下平・長谷川検定は安全であり,あやまった発見をする確率は小さいのですが,一方で本当の発見を見逃してしまう可能性が高いことが分かっています (false negativeの確率が大きい).つまりブートストラップ法と下平・長谷川検定はどちらも検定のバイアスがあり,その方向が異なっているのです.両者のちょうど間にバイアスがゼロ(つまり不偏)となる理想的な検定手法があることになります.Shimodaira (2002)によって開発されたマルチスケール・ブートストラップ法は,この理想的な検定を近似的に実現します.このアルゴリズムを実装したソフトウエア(Shimodaira and Hasegawa 2001,被引用回数)はISI New Hot Papersにも選ばれています.
計算した確率値を図4に示します.確率値が閾値0.05以上になる系統樹は青線で囲んだ6個あり,図2や図3と比べると,それらの間の結果になっています.現在真実と考えられている7番目の系統樹は確率値が0.10となって真実の可能性があると判断されています.これは図3でもいえたのですが,マルチスケール・ストラップ法のほうが下平・長谷川検定より真実と判断する系統樹の数を絞り込んでいて,より望ましいのです.
4. 参考資料
マルチスケール・ブートストラップ法の解説記事
高精度リサンプリング法
- 下平英寿.ブートストラップ法の幾何学とスケール変換 [PDF] [HTML] 2004-09 (日本数学会 特別講演)
- 日本数学会 特別講演 (2004/09/20)の講演スライド [PDF]
- H. Shimodaira (2004a). Approximately unbiased tests of regions using multistep-multiscale bootstrap resampling, Annals of Statistics, 32, 2616-2641. [PDF]
- H. Shimodaira (2004b). Technical details of the multistep-multiscale bootstrap resampling. Research Reports B-403. Department of Mathematical and Computing Sciences, Tokyo Institute of Technology, Tokyo, Japan. [PDF]
- H. Shimodaira and M. Hasegawa (2001). CONSEL: for assessing the confidence of phylogenetic tree selection, Bioinformatics, 17, 1246-1247. [PDF] [ソフトウエア]
- H. Shimodaira (2002). An approximately unbiased test of phylogenetic tree selection, Systematic Biology, 51, 492-508. [PDF]




